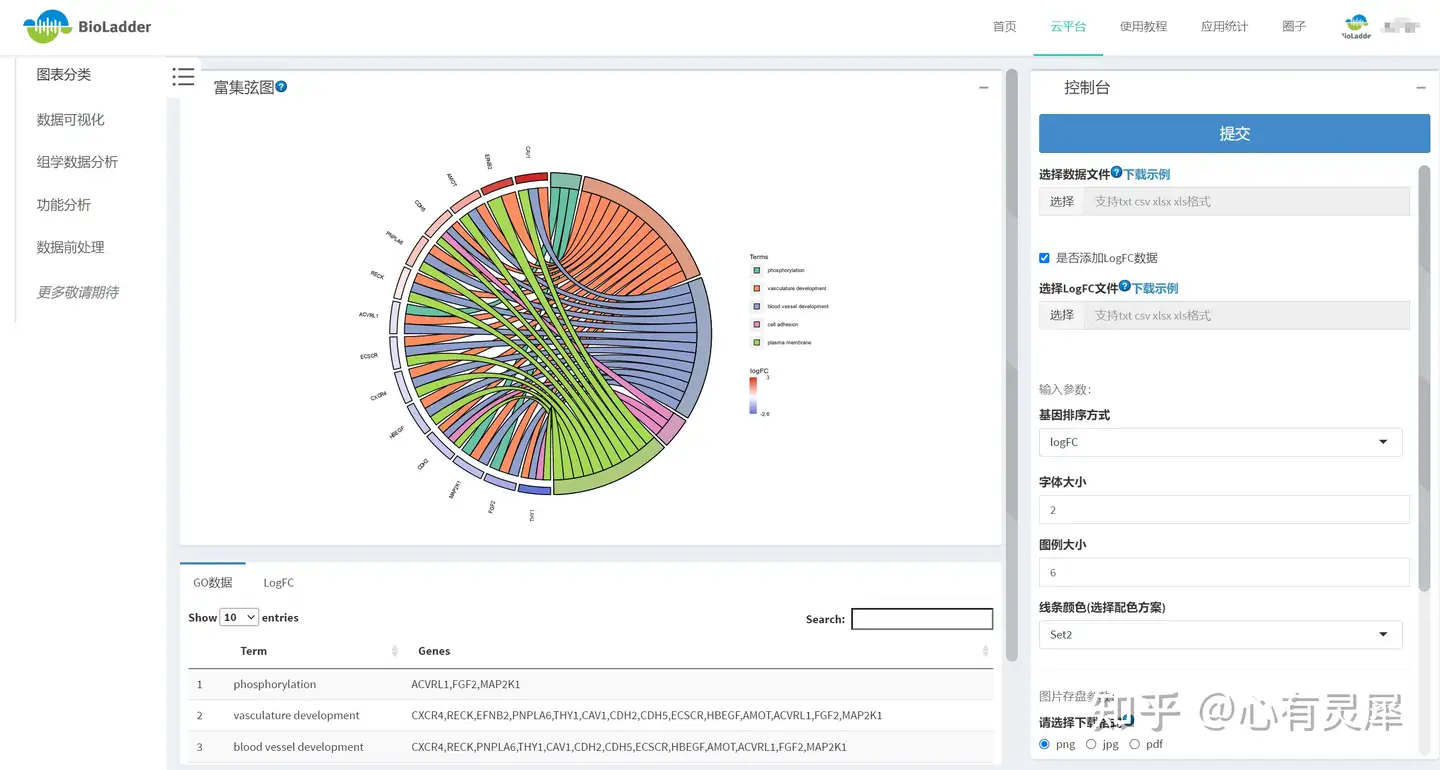
30 富集弦图
30.1 什么是富集弦图?
富集弦图:分别展示出每个Term富集到哪些基因以及这些基因的上下调情况。
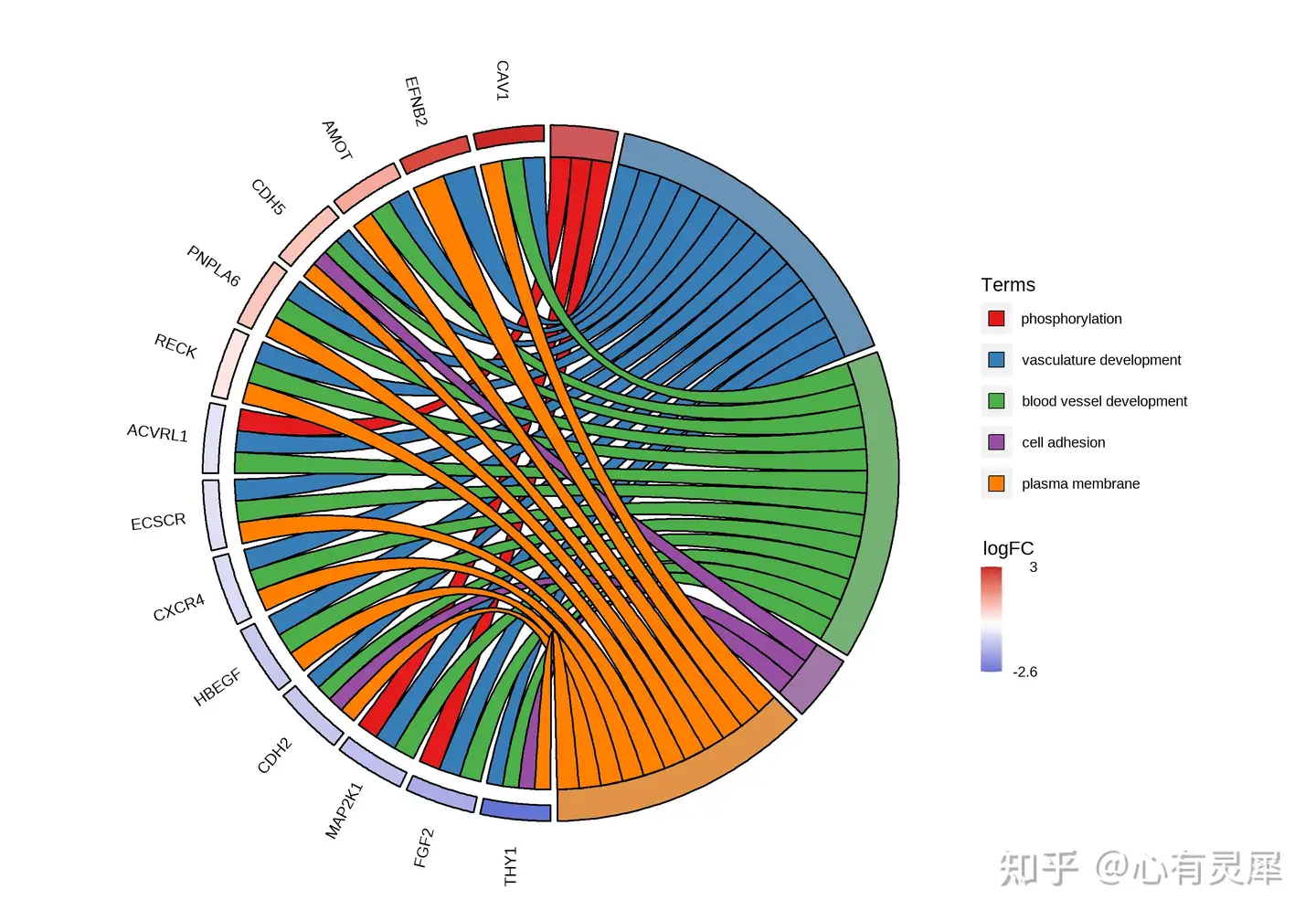
左侧为差异基因,颜色变化表示logFC从小到大的变化,红色为上调,蓝色为下调;右侧为富集结果的Term,两者相连表示基因富集到该Term中。
富集弦图一般显示3-10个通路较为合适,太多通路,会导致太过密集,反而看不清。
本文我们就来讨论一下富集弦图是如何绘制的。
30.1.1 绘图前的数据准备
需要2个文件
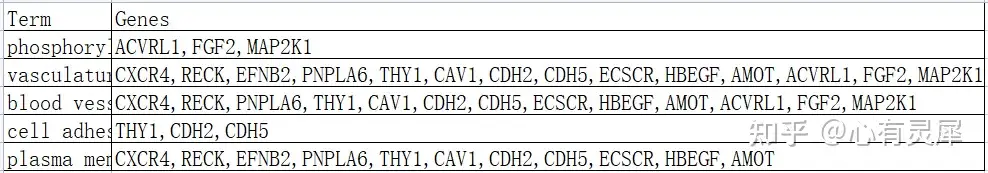
30.1.2 每个通路对应哪些基因
包含2列数据,第1列为通路名称,第2列为每个通路对应的基因,基因之间用”,“或”, “或”|“分隔开。
demo数据可以在https://www.bioladder.cn/shiny/zyp/bioladder2/model/bioladder1/GOplot/demoData1.txt下载。
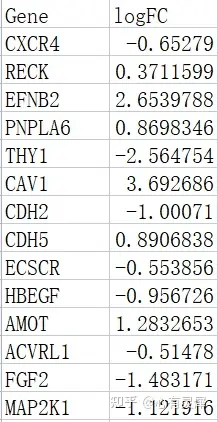
30.1.3 每个基因对应的logFC(可选)
第一列为基因名称,第二列为logFC。注意基因要和第一个文件对应。
demo数据可以在https://www.bioladder.cn/shiny/zyp/bioladder2/model/bioladder1/GOplot/demoData2.txt下载。
30.1.4 R语言怎么画富集弦图
# 转载所需要的包
library(tidyverse) # 用于数据处理
library(GOplot) # 用于绘图 安装方式 devtools::install_github("wencke/wencke.github.io")
library(RColorBrewer) # 颜色
# 读取数据
dfTerm = read.delim("https://www.bioladder.cn/shiny/zyp/bioladder2/model/bioladder1/GOplot/demoData1.txt")
dfFC = read.delim("https://www.bioladder.cn/shiny/zyp/bioladder2/model/bioladder1/GOplot/demoData2.txt")
# 整理数据
dfClean = dfTerm %>%
separate_rows(Genes,sep = ",") %>%
left_join(dfFC,by=c("Genes"="Gene"))
dfPlot = chord_dat(data.frame(dfClean), process=unique(dfClean$Term))
# 绘图
GOChord(dfPlot,
title="GOChord plot", # 标题名称
space = 0.02, # 基因方格之间的空隙
gene.order = "logFC", # 基因的排序方式,可以按照"logFC", "alphabetical", "none",
gene.space = 0.25, # 基因标签离图案的距离
gene.size = 5, # 基因标签的大小
lfc.col=c('firebrick3', 'white','royalblue3'), # logFC图例的颜色
ribbon.col=brewer.pal(ncol(dfPlot)-1, "Set2"), # 条带颜色设置
process.label = 8 # 图例标签的大小
)30.1.5 BioLadder生信云平台在线绘制富集弦图
不想写代码?可以用BioLadder生信云平台在线绘制富集弦图。
网址:
富集弦图-BioLadder生物信息在线分析可视化云平台www.bioladder.cn/web/#/chart/28