# A tibble: 603 × 1
IEAATDMTQKMADACQIYLDV
<chr>
1 NAKSFVGHAKTWSTIVSRSIG
2 GGGLQEVWSKFSDLPAGVNVT
3 DATLKWTRWIKADGATHLGVA
4 FGVTHVFYLNKVMKDALDWQD
5 EKQKLALANAKSFVGHAKTWS
6 GYWDSDMTERKTLFDVTTSLR
7 GTWSAADQLKVDAVSEFTATD
8 HSPFSTFSTMSSLSHALALAN
9 QVAFQLVVLRTDGTLAYLTSS
10 ATLKWTRWIKADGATHLGVAS
# ℹ 593 more rowsR语言如何绘制序列标识图seqlogo
什么是序列标识图seqlogo
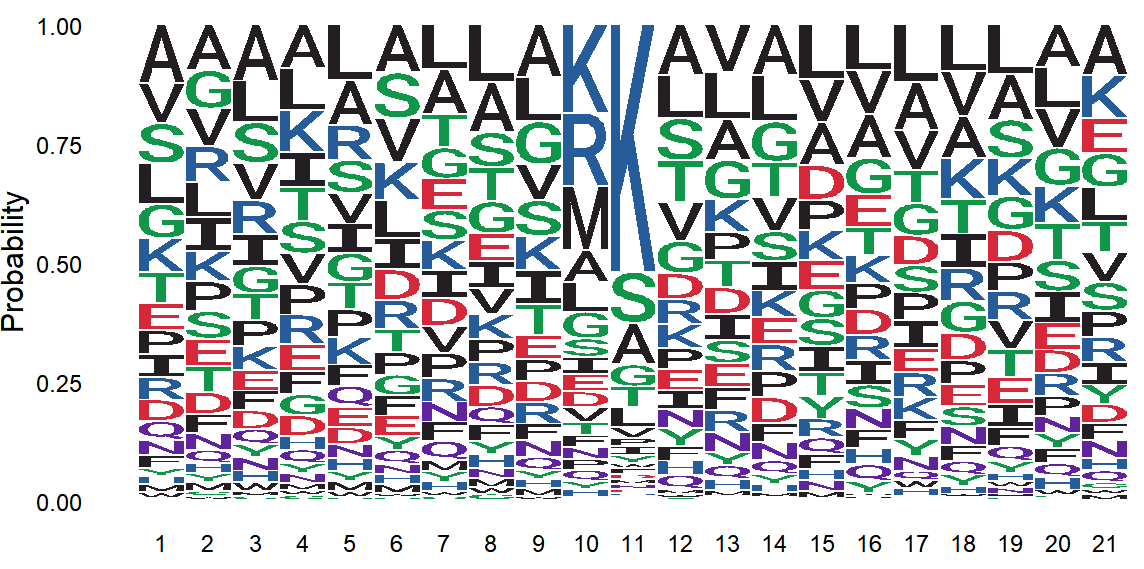
序列标识图(sequence logo),它是以图形的方式依次绘出序列比对中各个位置上出现的氨基酸或碱基,每个位置上氨基酸的累积可以反映出该位置上氨基酸的一致性。每个氨基酸对应图形字符的大小与氨基酸在该位置上出现的频率成正比。在生物信息分析中,经常需要做序列标识图。例如,突出序列比对中的保守位置,用于研究结构域序列相似性。
绘图前的数据准备
数据有且只有一列,没有行名与列名。内容是氨基酸序列。
demo数据可以从这下载:https://www.r2omics.cn/res/demodata/seqlogo.txt
R语言如何绘制序列标识图seqlogo
# 代码来源:https://www.r2omics.cn/
library(ggseqlogo)
# 读数据
df = read.delim("https://www.r2omics.cn/res/demodata/seqlogo.txt",header = F)
# 绘图
csl = c()
# # 可以自定义每个字母的颜色,例如
# csl <- make_col_scheme(chars = c("A"), cols = c("red"))
ggseqlogo(df,
method = "prob", # 定义统计方式"prob"百分比;bits;“custom”
col_scheme = csl) # 定义颜色方案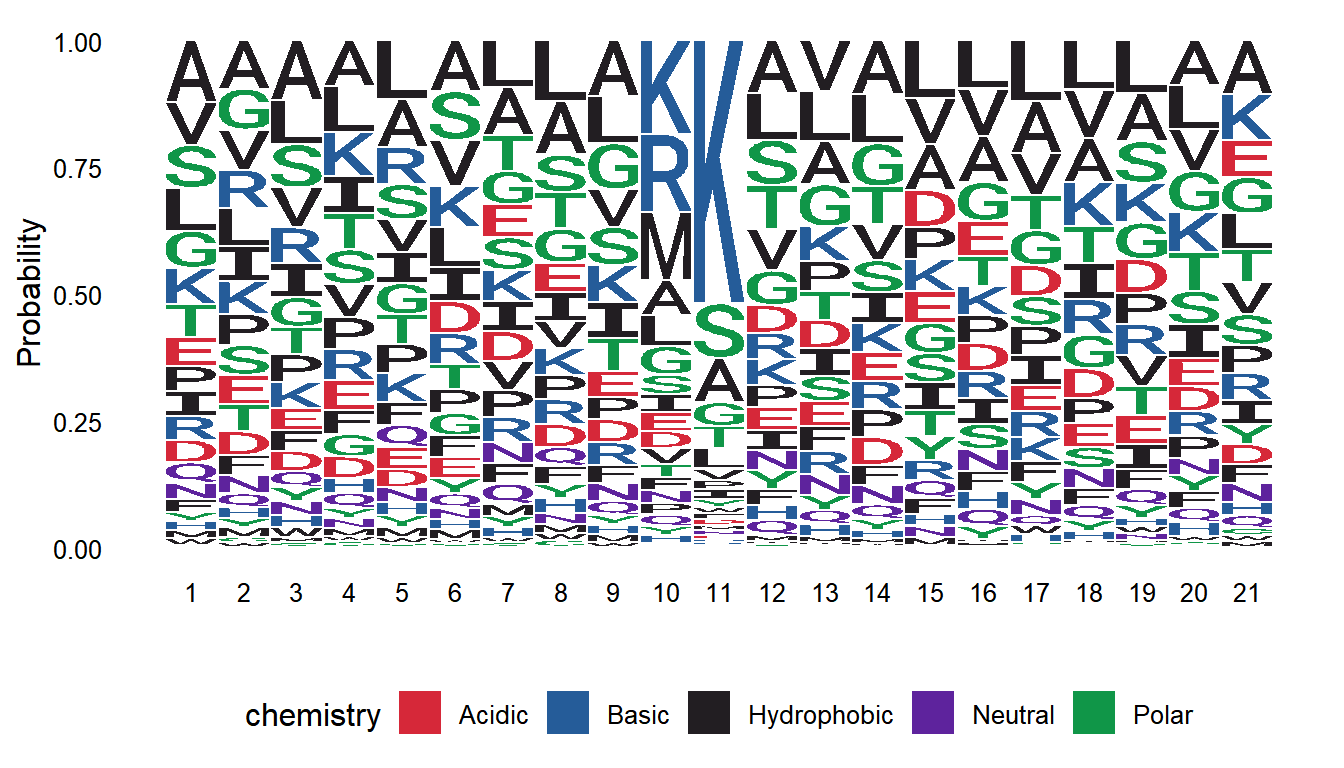
ggseqlogo是基于ggplot2开发的,所以还可以配合其他的ggplot2语法使用。