# A tibble: 2,319 × 2
Gene intensity
<chr> <dbl>
1 Gene1 26309304832
2 Gene2 6350142776
3 Gene3 5906849872
4 Gene4 3432857760
5 Gene5 2794144308
6 Gene6 2502199344
7 Gene7 2390313375
8 Gene8 2368543256
9 Gene9 1944088984
10 Gene10 1706848395
# ℹ 2,309 more rowsR语言如何绘制动态变化范围图
什么是动态变化范围图
蛋白定量值特征分布可以通过绘制动态变化范围图查看。
什么是动态变化范围图呢?对蛋白定量值从高到低排序后,绘制散点图。可以用来展示鉴定蛋白总数,定量检测数量级范围,以及定量值密集分布程度。
绘图前的数据准备
包含2列数据,第一列为蛋白基因等名称,第二列是定量值等数值信息。
demo数据可以从这下载:https://www.r2omics.cn/res/demodata/pointRank.txt
R语言如何绘制动态变化范围图
# 代码来源:https://www.r2omics.cn/
# 绘图思路为先排序,再绘制散点图
library(tidyverse)
# 读取数据
df = read.delim("https://www.r2omics.cn/res/demodata/pointRank.txt")
# 处理数据,排序
df = df %>%
arrange(desc(intensity)) %>%
mutate("x" = row_number())
# 绘图
ggplot(df,
aes(
x = x,
y = intensity
)
)+
geom_point(color="skyblue")+
theme_bw()+
labs(
x = "Ranked proteins",
y = "protein abundance"
)+
scale_y_continuous(breaks = 10^(-100:100), # 调整y轴的坐标轴样式为10^1,10^2...
labels = scales::trans_format("log10", scales::math_format(10^.x)),
trans="log10")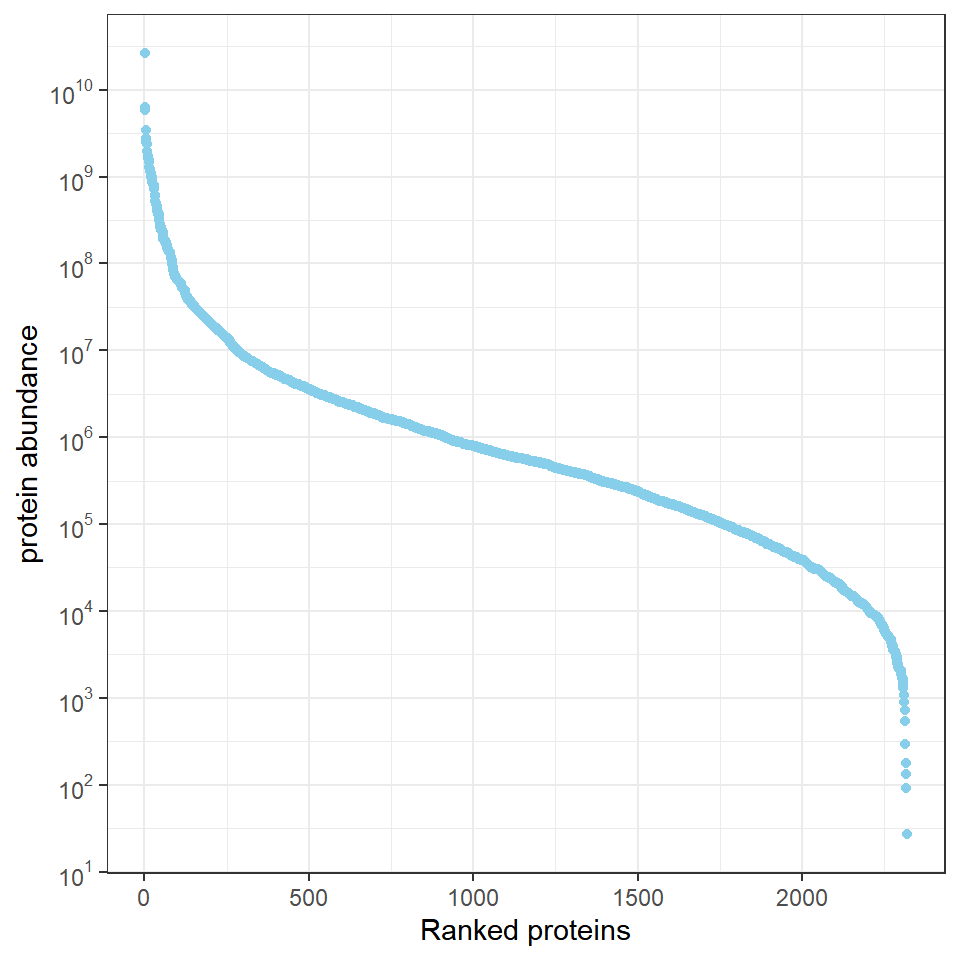
当然,还可以用geom_text()函数把目的蛋白标记在图上,看它们是低丰度蛋白还是高丰度蛋白。