# A tibble: 29 × 3
Gene X Y
<chr> <dbl> <dbl>
1 Gene1 18 19.8
2 Gene2 16.9 17.1
3 Gene3 6.34 8.64
4 Gene4 12.1 12.6
5 Gene5 15.5 16.8
6 Gene6 12.4 12.9
7 Gene7 24 25.1
8 Gene8 25.6 25.5
9 Gene9 29.0 28.9
10 Gene10 15.4 13.3
# ℹ 19 more rowsR语言如何绘制相关性散点图
什么是相关性散点图?
相关性是指两个或多个变量之间的关系或相互影响程度。若两组的值一起增大,我们称之为正相关,若一组的值增大时,另一组的值减小,我们称之为负相关。其值介于-1与1之间,即越接近1,越正相关;越接近-1,越负相关。
常见的有两种计算相关性的算法:皮尔逊相关性(pearson)和斯皮尔曼相关性(spearman)。皮尔逊相关性最常用,适合正态分布的数据。斯皮尔曼相关性是秩相关,不受极大极小值的影响。
在组学中的应用,例如,两个技术性重复实验的结果相关性很低,则说明数据有异常。
需要注意的地方,做样本之间的相关性的时候,蛋白之间要对应,不可随意打乱顺序。数据中有缺失值的情况,通常用”pairwise.complete.obs”算法处理缺失值,即两两配对删除缺失值。
相关系数散点图的关键是显示数据点的分布模式。如果数据点在图上呈现出一种明显的趋势,即呈线性关系,那么可以使用最小二乘回归线来进一步描述这种关系。相关系数则提供了一个度量两个变量之间线性相关性强弱的指标。
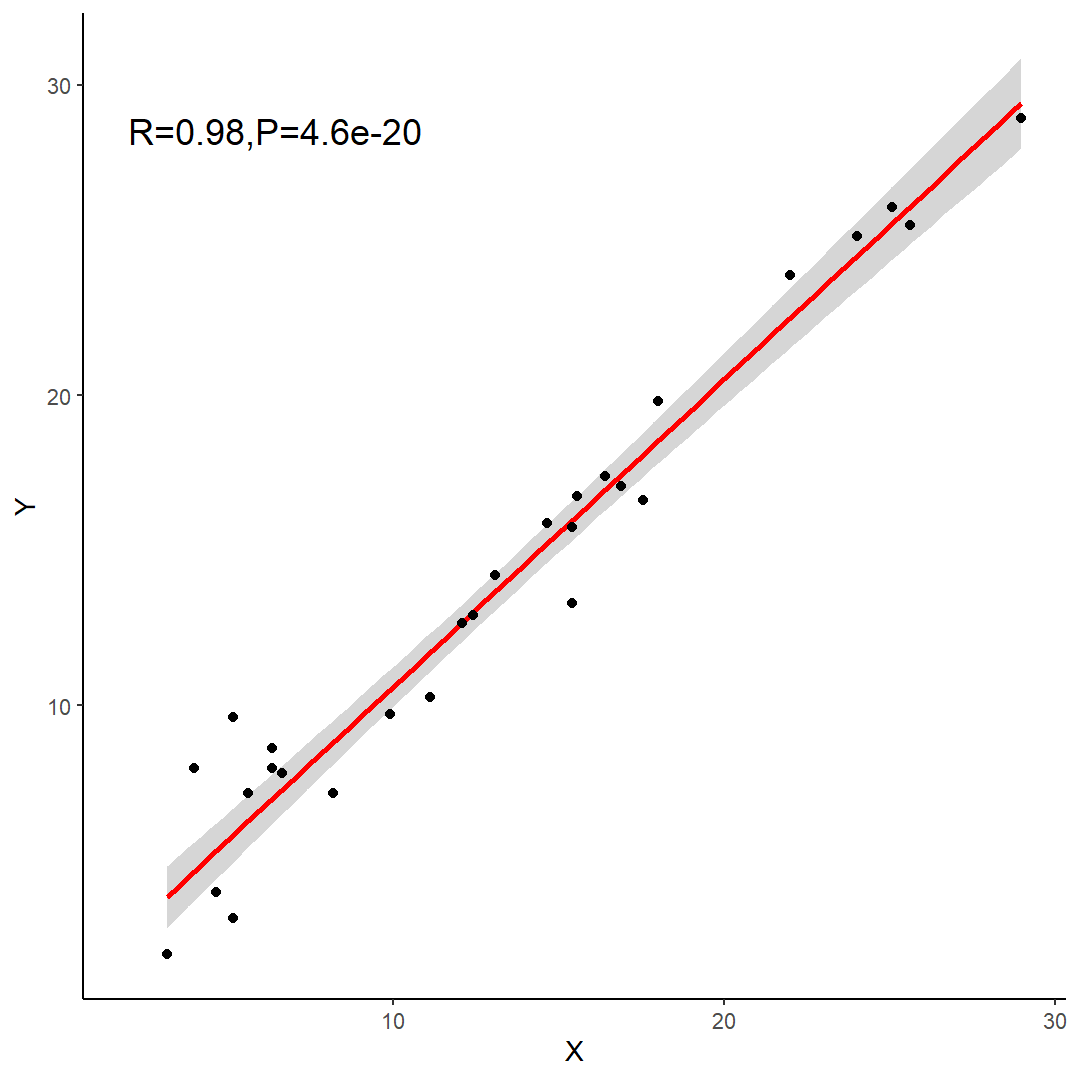
如图所示,红线是拟合曲线,灰色范围是置信区间,左上角的是相关性系数和对应的P值。
绘图前的数据准备
demo数据可以在这里下载https://www.r2omics.cn/res/demodata/corPoint.txt
R语言如何绘制相关系数散点图
# 代码来源:https://www.r2omics.cn/
library(ggplot2)
library(psych) # 提供corr.test函数,计算相关性和p值
# 读数据
df = read.delim("https://www.r2omics.cn/res/demodata/corPoint.txt", # 这里采用的网络上的数据,将此换为自己的数据
row.names = 1)
# 计算相关性
myCor = corr.test(df$X,df$Y,
use = "pairwise", # 缺失值处理的方式
method="pearson", # 计算相关性的方法有"pearson", "spearman", "kendall"
adjust = "none" # p值矫正的方法
)
# myCor$r # 查看相关性结果
# myCor$p # 查看p值
# 绘图
ggplot(df,aes(x=X,y=Y))+
geom_smooth(method="lm", # 拟合曲线
se=T, # 是否添加置信区间
color="red")+
geom_point()+ # 散点图
annotate(
"text", x = min(df,na.rm = T), y = max(df,na.rm = T), # 坐标位置,左上角
label = paste0("R=",round(myCor$r,2), # 注释标记内容相关性+P值
",P=",format(myCor$p, scientific = TRUE, digits = 2)), # P值采用科学计数法,保留2位小数
size = 5,hjust=0,vjust=1)+
theme_classic()