# A tibble: 32 x 11
mpg cyl disp hp drat wt qsec vs am gear carb
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
# i 22 more rowsR语言如何绘制相关性矩阵
什么是相关性矩阵?
相关性是指两个或多个变量之间的关系或相互影响程度。若两组的值一起增大,我们称之为正相关,若一组的值增大时,另一组的值减小,我们称之为负相关。其值介于-1与1之间,即越接近1,越正相关;越接近-1,越负相关。
常见的有两种计算相关性的算法:皮尔逊相关性(pearson)和斯皮尔曼相关性(spearman)。皮尔逊相关性最常用,适合正态分布的数据。斯皮尔曼相关性是秩相关,不受极大极小值的影响。
在组学中的应用,例如,两个技术性重复实验的结果相关性很低,则说明数据有异常。
需要注意的地方,做样本之间的相关性的时候,蛋白之间要对应,不可随意打乱顺序。数据中有缺失值的情况,通常用”pairwise.complete.obs”算法处理缺失值,即两两配对删除缺失值。
计算完相关性后,我们通过相关性矩阵做可视化。矩阵的上下中三个面板支持多种图案,有热力图,柱形图,散点图,折线图,饼图等多种模式可供选择。
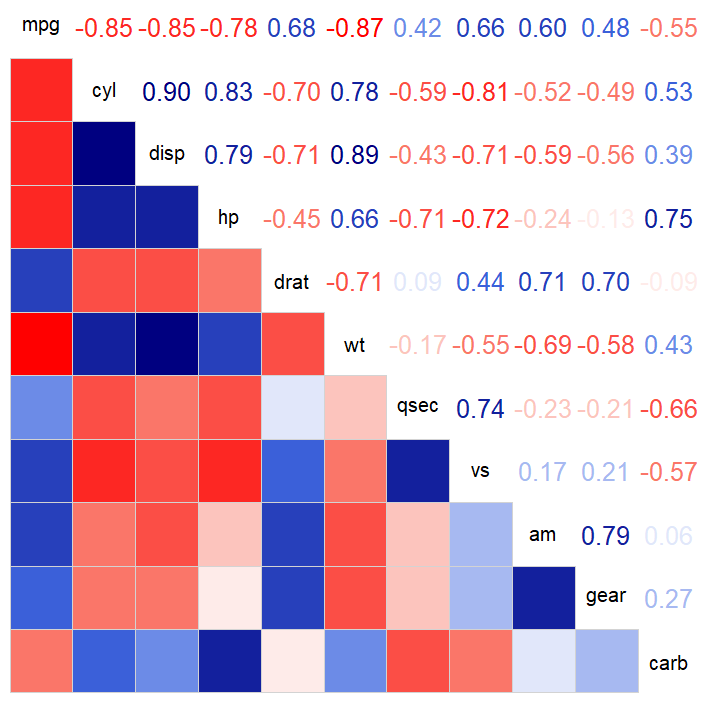
绘图前的数据准备
这里就用R语言自带的示例数据了,mtcars
R语言如何绘制相关性矩阵图
# 代码来源:https://www.r2omics.cn/
# 加载R包,没有安装请先安装 install.packages("包名")
library(corrgram)
# 读取相关性矩阵数据文件
df= mtcars
# 绘图
corrgram(df,
cex.labels = 1, # 样本文字大小
lower.panel="panel.pie", # 指定下方面板的形状,具体参数看下方注释
upper.panel="panel.cor", # 指定上方面板的形状,具体参数看下方注释
diag.panel=NULL, # 指定中间面板的形状,具体参数看下方注释
cor.method="pearson") # 计算相关性的方法有"pearson", "spearman", "kendall"
# 上、下面板可供选择的图形
# "散点" = "panel.pts"
# "柱形" = "panel.bar"
# "相关系数(带置信区间)"="panel.conf"
# "相关系数(带颜色)"="panel.cor"
# "ellipse"="panel.ellipse"
# "填充"="panel.fill"
# "饼图"="panel.pie"
# "阴影"="panel.shade"
# "空"=NULL
# 中间面板可供选择的图形
# "核密度图" = "panel.density"
# "最大最小值" = "panel.minmax"
# "空"=NULL