# A tibble: 228 x 3
Time Status Group
<int> <int> <chr>
1 306 2 男
2 455 2 男
3 1010 1 男
4 210 2 男
5 883 2 男
6 1022 1 男
7 310 2 女
8 361 2 女
9 218 2 男
10 166 2 男
# i 218 more rowsR语言如何做KM生存曲线
前言
本篇是R语言survival包和survminer包绘制KM生存曲线的教程。
什么是KM生存曲线?
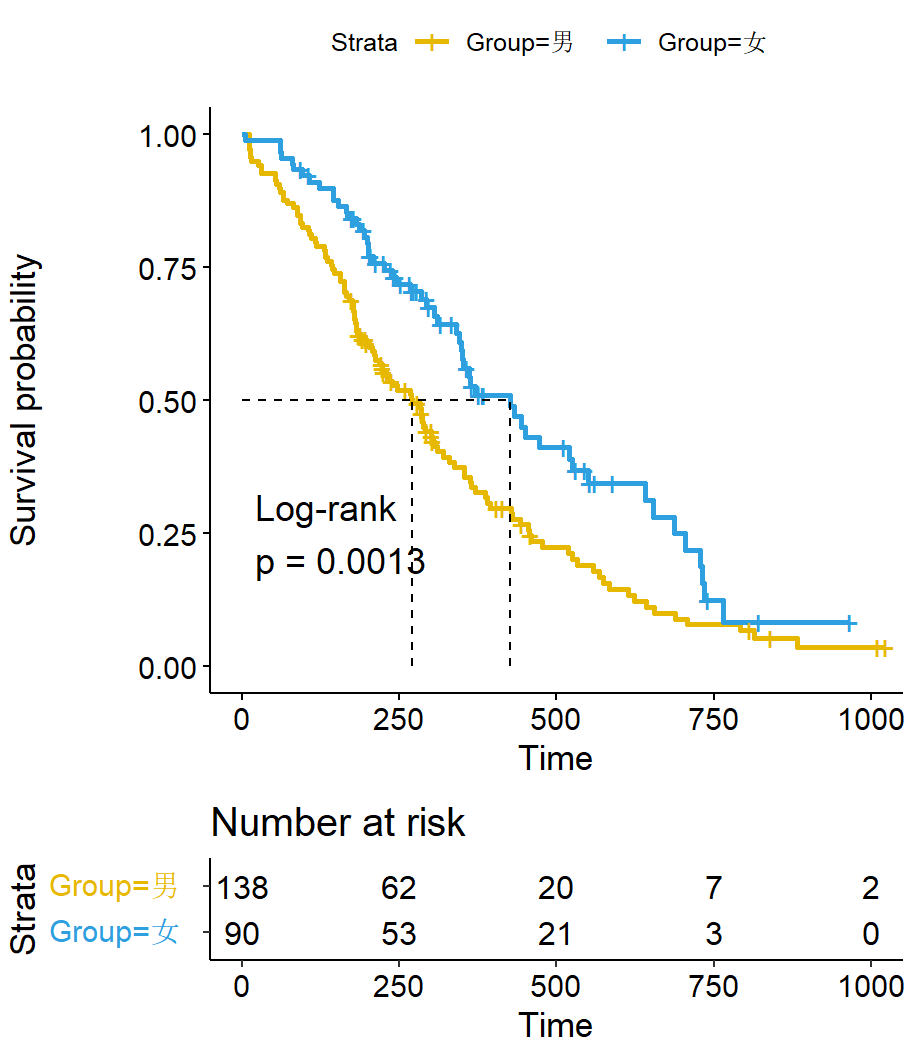
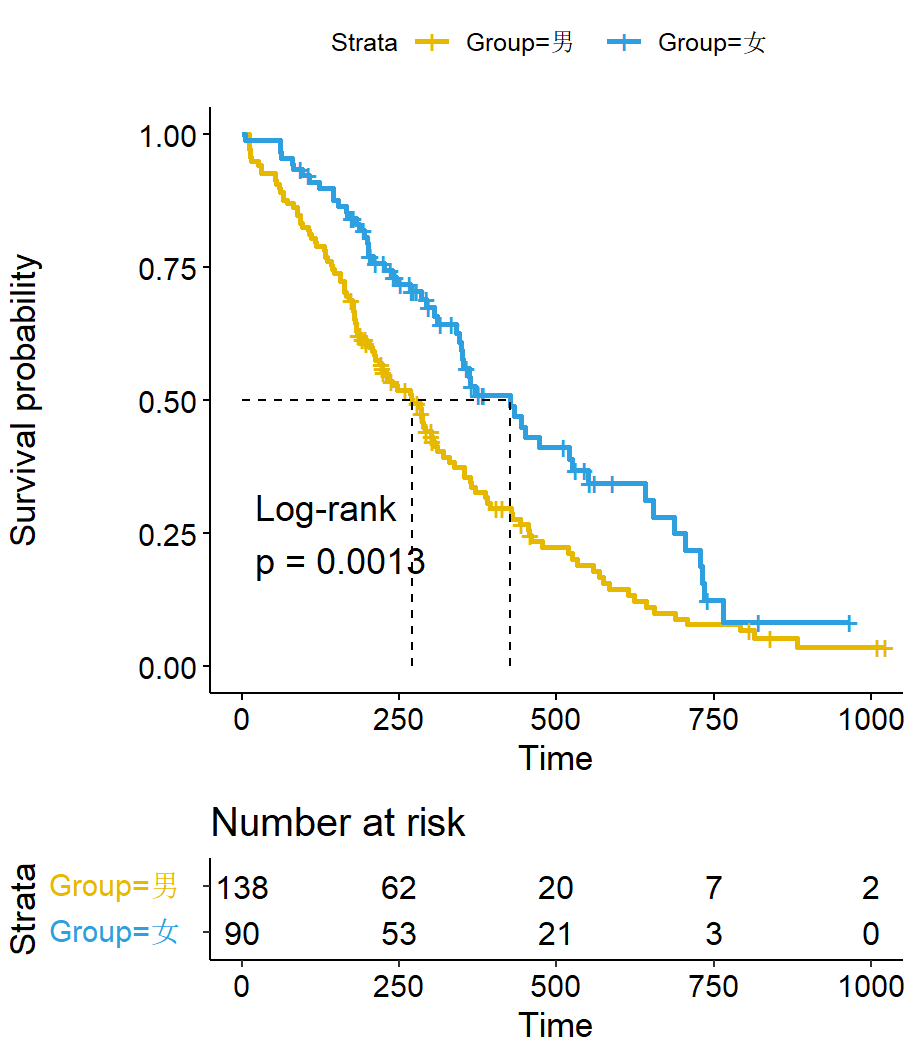
KM生存曲线(Kaplan-Meier survival curve)是用于描述在不同时间点上生存者的比例的一种统计工具。它常用于医学中对生存分析进行可视化和比较研究。
该曲线以时间为横轴,以生存率为纵轴,通过对样本生存时间数据进行分组并计算每组中生存者的比例,逐步绘制出曲线。该曲线能够精确反映出被观察对象在特定时间点上的存活率,并可以比较不同组别、不同治疗方案等因素对生存率的影响。
绘图前的数据准备
demo数据可以在https://www.r2omics.cn/res/demodata/KM.txt下载。
总共包含三列:
第1列是时间
第2列是状态:1表示观测结束(很多情况下,观测的结束可能是由于研究结束或者失访等原因),2表示死亡
第3列是:分组
R语言如何绘制KM生存曲线
# 代码来源:https://www.r2omics.cn/
# 加载包
library(survival)
library(survminer)
# 加载数据
df = read.delim("https://www.r2omics.cn/res/demodata/KM.txt")
# 根据时间和状态构建生存分析模型
fit = survfit(Surv(Time, Status) ~ Group, data = df)
# 绘图
ggsurvplot(
fit, # 生存分析模型对象
data = df, # 数据框
palette = c("#E7B800", "#2E9FDF"), # 设置颜色
conf.int = FALSE, # 是否显示置信区间
pval = T, # 是否显示P值
pval.method = T, # 显示P值的计算方法
surv.median.line = "hv", # 是否显示中位数生存时间曲线,hv表示显示,none不显示
risk.table = T, # 是否显示风险表格
cumevents = F, # 是否显示累积死亡表格
cumcensor = F, # 是否显示累积截尾censored表格
)