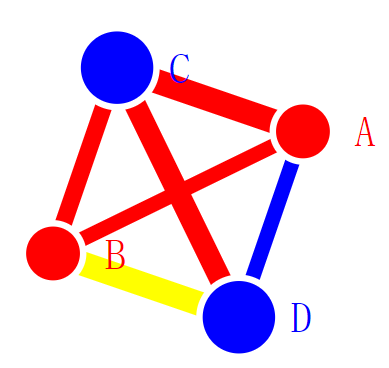
# A tibble: 4 × 4
name group size group_color
<chr> <chr> <int> <chr>
1 A group1 1 red
2 B group1 1 red
3 C group2 10 blue
4 D group2 10 blue R语言如何绘制相互作用网络图
前言
本篇是使用R语言的networkD3包绘制可交互网络图的教程。
什么是相互作用网络图?
要素:结点,连线和布局。
在相互作用网络图中,结点通常代表个体或实体,连线则表示它们之间的相互作用或联系。
绘图前的数据准备
结点数据
包含4列,第一列是边数据文件中的节点,第二列是该节点所在的分组。第三列定义节点的大小。第四列定义分组的颜色。
demo数据可以在这下载:https://www.r2omics.cn/res/demodata/networkD3/node.txt
连线数据
包含4列,前两列定义连线,从哪个节点到哪个节点。第三列是数值,定义连线粗细。第四列定义连线的颜色。
demo数据可以在这下载:https://www.r2omics.cn/res/demodata/networkD3/link.txt
# A tibble: 6 × 4
source target value colour
<chr> <chr> <int> <chr>
1 A B 12 red
2 A D 12 blue
3 A C 32 red
4 B C 16 red
5 B D 32 yellow
6 D C 26 red R语言如何绘制相互作用网络图
第一步读取了结点和分组数据。然后对结点的出现顺序排了个序。在圆形布局时,设置文本标记旋转到合适的角度。最后转换为mygraph图形数据格式。
# 代码来源:https://www.r2omics.cn/
library(tidyverse)
library(networkD3)
# 读取连线文件和节点文件
MisLinks = read.delim("https://www.r2omics.cn/res/demodata/networkD3/link.txt")
MisNodes = read.delim("https://www.r2omics.cn/res/demodata/networkD3/node.txt")
# 处理数据
# 因为networkD3需要的连线数据,是节点文件里的名称的索引。所以,需要做一个名称到索引的转化
Node2index = list()
Node2index[MisNodes$name] = 0:length(MisNodes$name)
MisLinks = MisLinks %>%
mutate(source2 = unlist(Node2index[source])) %>%
mutate(target2 = unlist(Node2index[target]))
# 定义颜色
group2project = paste(unique(MisNodes$group),collapse = '","')
color2project = paste(unique(MisNodes$group_color),collapse = '","')
my_color <- paste0('d3.scaleOrdinal().domain(["',group2project,'"]).range(["',color2project,'"])')
# 绘图
fig = forceNetwork(Links = MisLinks,
Nodes = MisNodes,
Source = "source2",
Target = "target2",
Value ="value",
NodeID = "name",
Group = "group",
opacity= 1, # 透明度
Nodesize="size",
zoom = TRUE, # 是否可以缩放
opacityNoHover=1, # 鼠标没有悬浮在节点上时,文字的透明度(0-1)
colourScale = JS(my_color), # 节点颜色,JavaScript
legend=T,
fontSize = 10,
linkColour= MisLinks$colour
)
# 保存为网页
# htmlwidgets::saveWidget(fig,"network.html")