# A tibble: 200 × 5
id A B C D
<chr> <dbl> <dbl> <dbl> <dbl>
1 ID1 7.29 7.29 7.29 7.29
2 ID2 8.90 8.90 8.90 8.90
3 ID3 5.56 5.56 5.56 5.56
4 ID4 6.21 6.21 6.21 6.21
5 ID5 2.75 2.75 2.75 2.75
6 ID6 4.67 4.67 4.67 4.67
7 ID7 5.14 5.14 5.14 5.14
8 ID8 5.45 5.45 5.45 5.45
9 ID9 8.47 8.47 8.47 8.47
10 ID10 6.70 6.70 6.70 6.70
# ℹ 190 more rowsR语言如何绘制抖动散点图
抖动散点图采用随机抖动的方法,将数据点沿着其x轴坐标随机移动一定距离,以避免数据点之间的重叠。由于抖动的强度是随机的,因此不同的数据集可能会产生不同的效果。
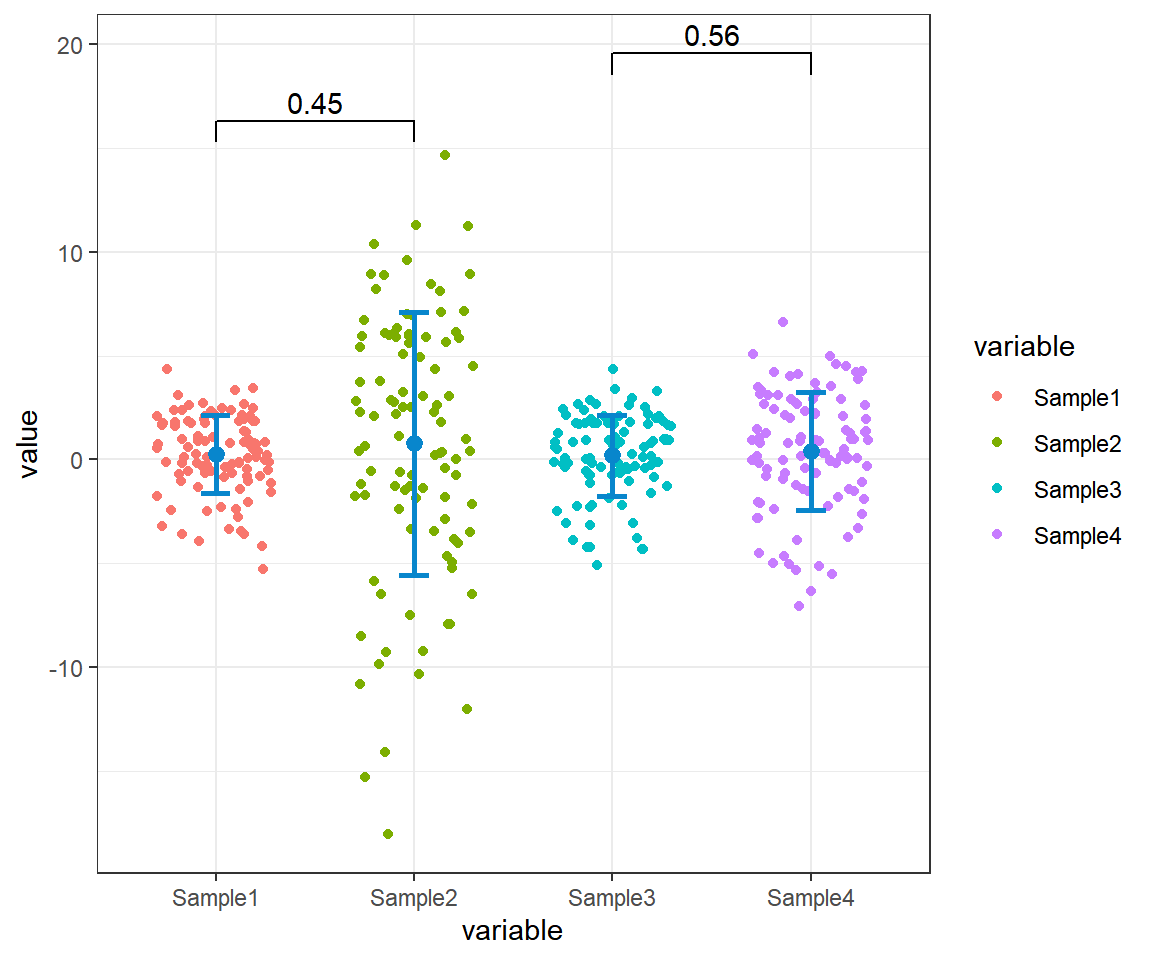
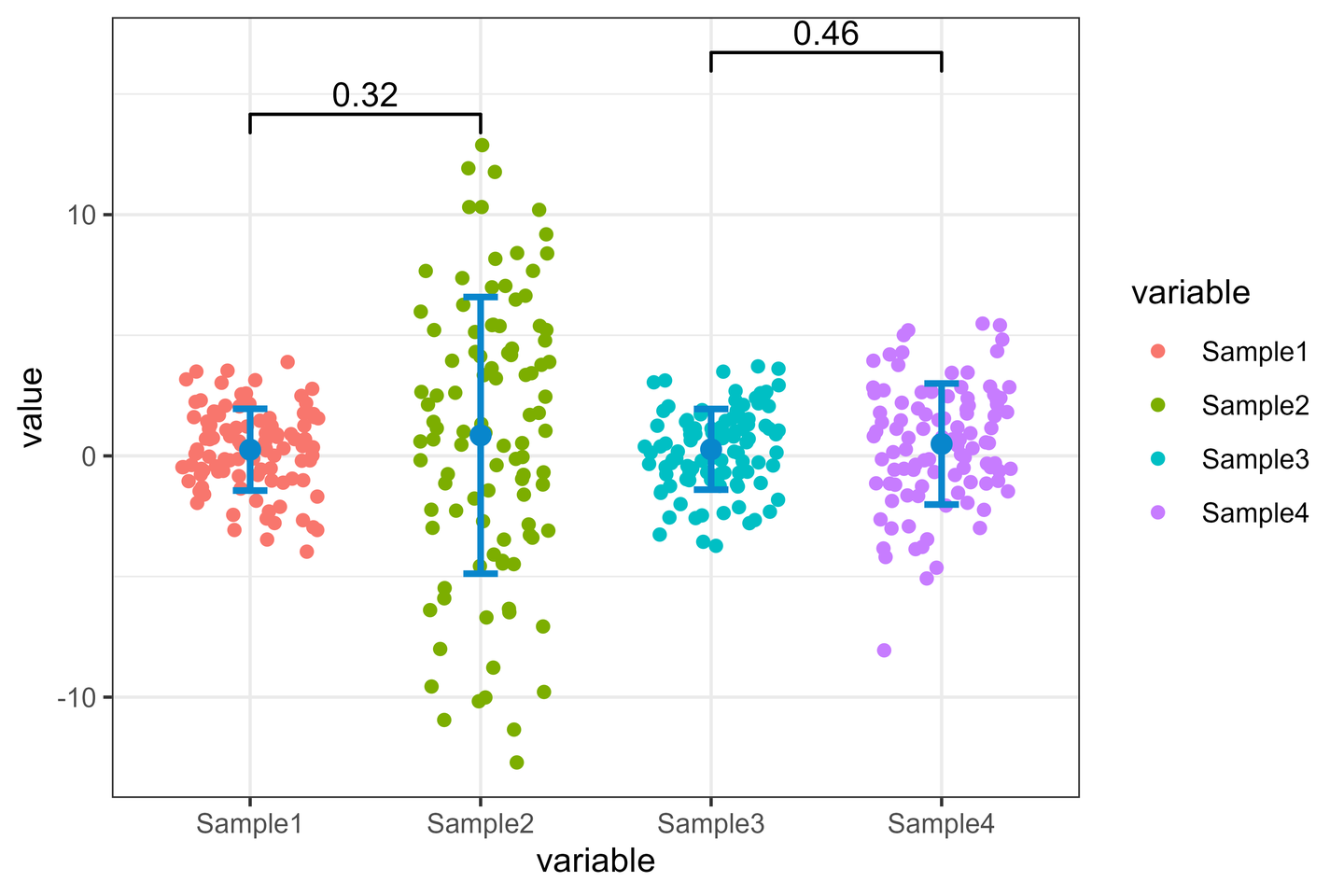
本教程我们还会在图上添加误差线和显著性标记。
什么是抖动散点图?
绘图前的数据准备
demo数据可以在https://www.r2omics.cn/res/demodata/beeswarm.txt下载。
第一列为名称列,其他列为数据列。
如果是转录组蛋白组等生物学数据,则每一列代表一个样本,每一行代表一个基因。
R语言如何绘制抖动散点图
# 代码来源:https://www.r2omics.cn/
# 加载R包,没有安装请先安装 install.packages("包名")
library(tidyverse)
library(ggsignif)
# 读取数据文件
df = read.delim("https://www.r2omics.cn/res/demodata/boxplot.txt"# 这里读取了网络上的demo数据,将此处换成你自己电脑里的文件
)[1:100,]
# 把数据转换成ggplot常用的类型(长数据)
df = df %>%
pivot_longer(-1,names_to = "variable",values_to = "value")
# 绘图
ggplot(df,aes(x=variable,y=value))+
geom_jitter(aes(color=variable),width = 0.3,height = 0)+ # 抖动散点图,width = 0.3,height = 0只左右抖动,不上下抖动
stat_summary(geom = 'pointrange', # 加中间那个点(平均值)
color='#0886CC',
fun = mean
)+
stat_summary(geom = 'errorbar', # 加上下误差线,(平均值加减标准差)
color='#0886CC',cex=1,width=0.15,
fun = mean,
fun.max = function(x) mean(x) + sd(x),
fun.min = function(x) mean(x) - sd(x)
)+
theme_bw()+
geom_signif( # 添加显著性标签
comparisons=list(c("Sample1","Sample2"),c("Sample3","Sample4")), # 选择你想在哪2组上添加标签
step_increase = 0.1,
test="t.test", # "t 检验,比较两组(参数)" = "t.test","Wilcoxon 符号秩检验,比较两组(非参数)" = "wilcox.test"
test.args = list("var.equal" = T), # 等方差
map_signif_level=F # 标签样式F为数字,T为*号
)