# A tibble: 200 × 5
id A B C D
<chr> <dbl> <dbl> <dbl> <dbl>
1 ID1 7.29 7.29 7.29 7.29
2 ID2 8.90 8.90 8.90 8.90
3 ID3 5.56 5.56 5.56 5.56
4 ID4 6.21 6.21 6.21 6.21
5 ID5 2.75 2.75 2.75 2.75
6 ID6 4.67 4.67 4.67 4.67
7 ID7 5.14 5.14 5.14 5.14
8 ID8 5.45 5.45 5.45 5.45
9 ID9 8.47 8.47 8.47 8.47
10 ID10 6.70 6.70 6.70 6.70
# ℹ 190 more rowsR语言如何绘制蜂群图
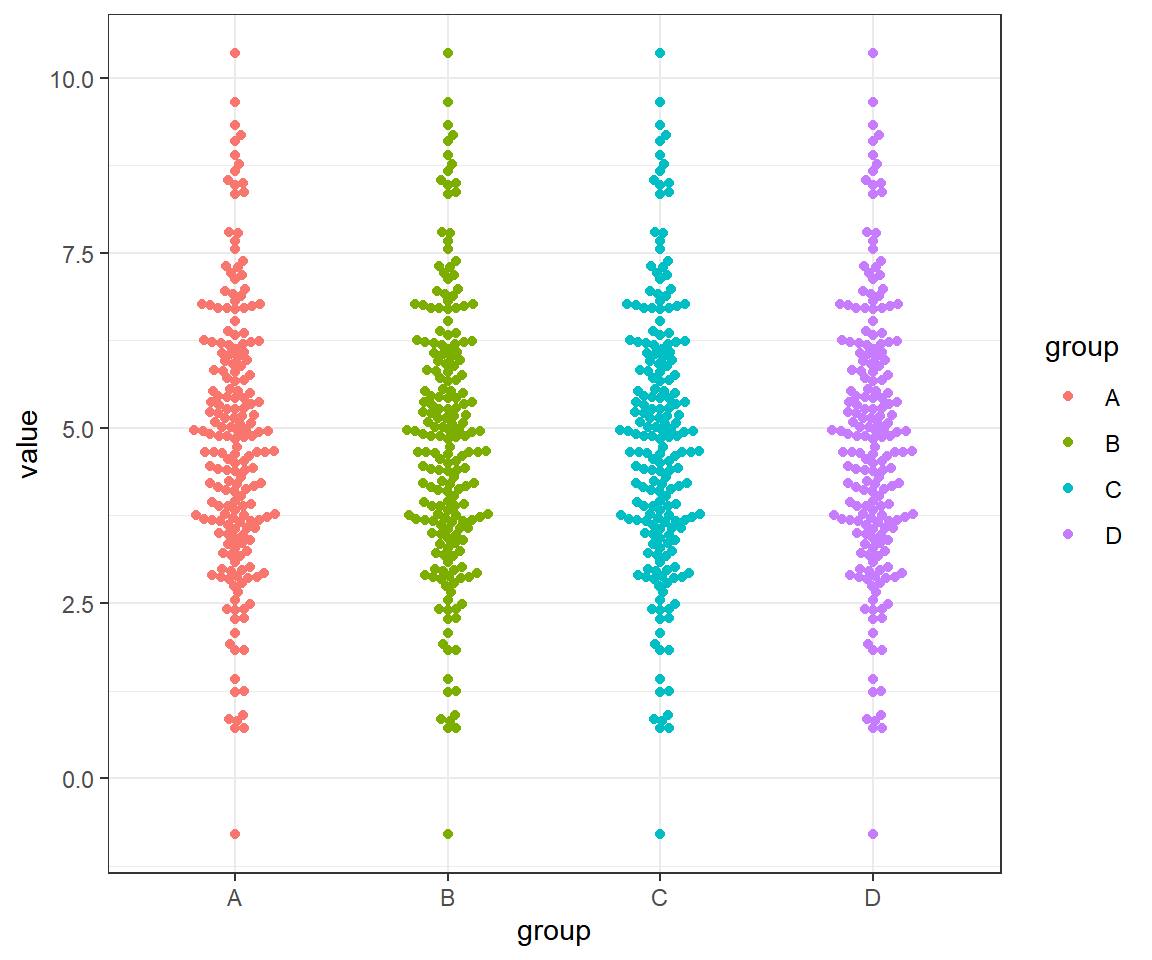
什么是蜂群图?
蜂群图(beeswarm plot)是一种可视化离散数据分布的图形。它沿着 X 轴类别标签中心线向两侧,同时逐步向上均匀而对称的展开,避免了数据点之间的重叠,使得数据的密度和分布更容易观察和理解。
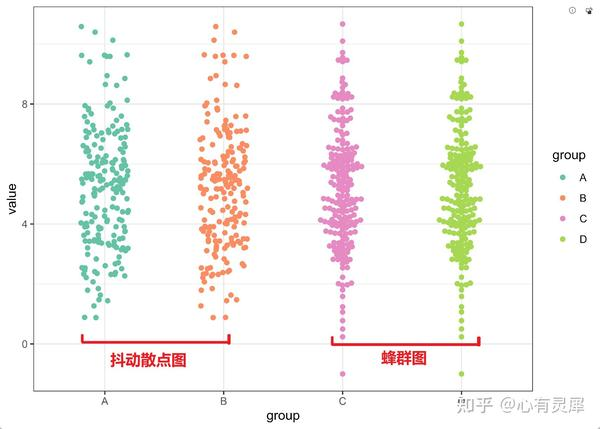
与抖动散点图相比的区别
抖动散点图采用随机抖动的方法,将数据点沿着其x轴坐标随机移动一定距离,以避免数据点之间的重叠。由于抖动的强度是随机的,因此不同的数据集可能会产生不同的效果。
与抖动散点图相比,蜂群图是紧密不重叠的,方便读者观察数据的分布规律,但需要设计者进行适当的布局设计,使图形更加美观和易读。
绘图前的数据准备
demo数据可以在https://www.r2omics.cn/res/demodata/beeswarm.txt下载。
第一列为名称列,其他列为数据列。
如果是转录组蛋白组等生物学数据,则每一列代表一个样本,每一行代表一个基因。
R语言如何绘制蜂群图
# 代码来源:https://www.r2omics.cn/
# install.packages('ggbeeswarm')
library(tidyverse)
library(ggbeeswarm) # 提供geom_beeswarm函数,用于绘制蜂群图
# 读数据
# 这里读取了网络上的demo数据,将此处换成你自己电脑里的文件
df = read.delim("https://www.r2omics.cn/res/demodata/beeswarm.txt")
# 把数据转换成ggplot需要的数据类型(长数据)
dfPlot = df %>%
pivot_longer(-1,names_to = "group",values_to = "value")
# 绘图
ggplot(dfPlot,aes(x=group,y=value,color=group))+
geom_beeswarm(method = "swarm")+
theme_bw()